


引言
第三代CRISPR基因敲除试剂盒(Cas9x3.0)是海星生物在多年基因编辑经验的基础上研发的高效快捷的基因敲除试剂盒。试剂盒产品函括了人(Human)和小鼠(Mouse)的所有基因。
Cas9x3.0试剂盒包含2个经算法或实验优化的gRNA序列载体和特异性的同源DNA片段,DNA片段含有抗性(PuroR)和GFP荧光,并携带有一段特殊同源序列,这个插入的同源片段不仅为后续基因敲除细胞的筛选提供药物抗性,也可以通过GFP流式筛选获得敲除细胞。
第三代的Cas9x3.0试剂盒对分裂细胞和非分裂细胞都有非常高的敲除效率,特别适合于增殖正常,Lipo转染或电转效率较高且能建立单克隆的细胞株;原代细胞和增殖缓慢细胞需要预先进行转染效率优化,确定EG转染率不低于50%。
试剂盒构成:

推荐次数:
20 次
原理说明:
第三代CRISPR基因敲除试剂盒 (Cas9x3.0) 依据同源重组(Homology directed repair, HDR)和非同源性末端接合(Non-homologous end joining,NHEJ)修复原理进行产品研发,利用插入的修复原理实现目的基因的敲除。
特定优化的gRNA引导Cas9蛋白复合体Ribonucleoprotein(RNP)对特定基因位点进行切割,产生DNA双链断裂(Double-Strand Break, DSB),并诱导携带有特定同源序列的线性化DNA片段(该片段携带抗性基因和荧光基因)整合到DSB位点,再通过抗性(Puro)或荧光(GFP)进行筛选,从而获得携带有插入片段的基因敲除的细胞。
CRISPR基因敲除试剂盒 (Cas9x3.0) 的敲除效率是传统单gRNA基因敲除策略的2~5倍。因为其独有的敲除策略是通过在基因位导入同源的Donor DNA( Puro & GFP )从而达到敲除目的蛋白效果,同时也可以在DSB位点导入小片段的缺失导致序列移码,从而实现蛋白翻译提前终止。同时这两个原理的叠加,可以轻松获得多个“等位基因”完全敲除细胞株
原理图:

图 1 第三代CRISPR基因敲除试剂盒(Cas9x3.0)原理
操作流程:
第三代CRISPR基因敲除试剂盒 (Cas9x3.0) 配有eGFP对照载体,供实验过程摸索转染条件使用,可以按照使用的方法和细胞情况,进行实验条件摸索,建议48小时后的EG载体转染目的细胞的eGFP的阳性率不低于50%,Cas9x3.0试剂盒才能获得最佳敲除效果。基因敲除试剂盒 (Cas9x3.0)适用于“脂质体转染法”和“电转化法”。
1. 细胞准备
细胞复苏培养,使用增殖速度快,能够形成良好单克隆的细胞、细胞转染效率高的细胞是实验成功的关键因素,建议满足以下条件:
A. 复苏细胞要传代3次以上;
B. 尽可能使用克隆形成良好的细胞;
C. 转染前2天对细胞进行传代;
D. 转染前单天更换细胞培养基;
E. 转染前保持细胞在汇合度50~85%,使细胞处于最佳增殖状态。

图 2 细胞敲除技术流程与周期
2. 细胞转染
2.1 CRISPR基因敲除试剂盒 (Cas9x3.0)操作步骤(脂质体转染法)
本试剂盒推荐采用转染试剂是Lipofectamine 2000 Reagent (cat#11668-030),具体参照转染试剂说明材料。不同类型的转染试剂,请按照制造商的操作说明进行转染。 注意:悬浮细胞不推荐使用脂质体转染法。 |
本试剂盒以HEK293细胞24孔板转染为案例进行说明,采用的转染试剂是Lipofectamine® 2000 Reagent (cat#11668-030)。不同转染规格,可以根据不同培养皿的相对表面积的比例配置转染试剂(表1)。
表 1 Lipofectamine® 2000 Reagent转染规格说明

2.2 GFP转染测试(EG细胞转染率测试):
Lipofectamine 2000转染试剂的转染步骤(24孔板) ,293细胞进行GFP转染:
A. 转染前一天,胰蛋白酶消化细胞并计数(0.5-2×10^5),铺板细胞,使转染当天的密度为80-85%,转染准备前换液成为0.5ml Opti-MEM培养基。(不建议细胞过密进行转染)
B. 准备1.5mL EP管,编号管1,将0.5μg eGFP表达载体(EG管)溶于50ul Opti-MEM®培养基中混匀。
C. 准备1.5mL EP管,编号管2,将2ul lipo2000溶于50ul Opti-MEM®培养基中混匀。
D. 将管1全部转移到管2中,混匀,室温放置20min。
E. 将上述混合物加入步骤1中的培养细胞中,轻柔地来回晃动培养皿,使混合物均匀分布。
F. 将包含转染复合物和细胞的平板放在组织培养箱(37ºC, 5% CO2)中培养过夜后换成正常的培养基。
G. 培养48小时后观察细胞GFP的表达率,建议GFP阳性率大于50%。
H. 如果GFP的阳性率过低,需要对细胞状态、DNA的转染量(0.5~1ug/24孔)等进行优化。
注意:转染的过程使用的所有的培养基均为无抗生素培养基,否则将导致细胞死亡。由于EG载体使用的CAG驱动,能够保证较高的表达效率。

图 3 293细胞转染GFP的情况/EG对照质粒
2.3 gRNA:Donor转染实验(正式实验):
按照对应的转染试剂操作说明,在完全培养基中分别对2个gRNA/Donor进行转染:
表 2 细胞敲除实验组


图 4 293细胞转染分组示意图
Lipofectamine 2000转染试剂的转染步骤(24孔板) ,293细胞进行敲除载体转染:
A. 转染前一天,胰蛋白酶消化细胞并计数(0.5-2×10^5),铺板细胞24孔中的4个孔,使转染当天的密度为70-85%,转染准备前换液成为0.5ml Opti-MEM培养基。(不建议细胞过密进行转染)
B. 准备1.5mL EP管,编号管1~4,加入50 ul Opti-MEM培养基,将G1-DR敲除组、G2-DR敲除组、eGFP对照组的DNA分别加入1~3到50ul Opti-MEM培养基中混匀。
C. 准备1.5mL EP管,编号管A~D,加入50 ul Opti-MEM培养基,并将2ul lipo2000加入到Opti-MEM培养基中轻柔混匀。
D. 将管1~4分别转移到管A~D中,混匀后,室温放置5 min。
E. 将上述混合物加入步骤A中的培养细胞中,轻柔地来回晃动培养皿,使混合物均匀分布,并做好标记,记录时间。
F. 将包含转染复合物和细胞的平板放在组织培养箱(37ºC, 5% CO2)中培养过夜后,换成正常的完全培养基。
G. 培养48小时后观察G1/DR、G2/DR细胞GFP的表达率(20~80%),EG组GFP阳性率应大于50%。
H. 如果GFP效率符合预期,即可以进入到后续的传代或筛选的步骤。
I . 如果G1/DR、G2/DR细胞GFP的荧光率在20~40%建议在转染后的48小时进行药物puro药物筛选;G1/DR、G2/DR荧光率在41~80%的可以按照传代后再进行药物筛选。(具体实验表面,电转后48小时开始药筛,对于GFP荧光率低的细胞具有更好的效果)
注意:
1)转染的过程使用的所有的培养基均为无抗生素培养基,否则将导致细胞死亡。
2)由于G1/G2载体使用的EF1a驱动GFP:P2A:Puro (如果增殖速度慢的细胞会出现GFP不强的情况)
3)如果G1/DR、G2/DR荧光率低于20%,建议重新优化转染实验条件。
2.2 CRISPR基因敲除试剂盒 (Cas9x3.0)操作步骤(电转化法)
本试剂盒推荐Neon转染系统,具体参照设备对每种细胞参数。不同类型的电转设备,请按照制造商的操作说明进行优化。
注意:悬浮细胞推荐使用电转化法。
本试剂盒以悬浮细胞K562为案例进行电转化法操作说明,采用的电转仪是Neon (Cat# MPK5000)。

图 5 K562细胞电转示意图
细胞电转染步骤(24孔板) ,K562细胞进行敲除载体转染:
A. K562细胞电转实验使用仪器为 Neon™ 转染仪(Cat# MPK5000),参考官网参数 1450V, 10ms, 3pulses,使用10 μL T Buffer 体系,电转杯缓冲液为 3 mL E Buffer。
B. 向六孔板内加入500ul培养基,放置于培养箱备用。
C. 将0.5 μg gRNA和0.5 μg Donor DNA分别加入 T Buffer中混匀,补齐T Buffer至10 μL(为了保证实验顺利,可以适当增加2ul的T Buffer);
D. 将细胞离心后使用PBS重悬,计数取 1×105cells 至 EP 管内,离心后使用 步骤C中10 μL T Buffer 重悬细胞,轻柔吹打混匀。
E. 在 Neon™ 转染仪界面输入参数 1450 V, 10 ms, 3 pulses,用 Neon™ 移液器吸取 10 μL 细胞悬液。
F. 将 Neon™ 移液器置于电转杯中,点击界面“Start”进行电转。
G. 将电转后的细胞轻柔加入24孔板培养基中,放置培养箱培养48小时,便可以观察细胞的GFP表达按情况。
H. 培养48小时后观察G1/DR、G2/DR细胞GFP的表达率(20~80%),EG组GFP阳性率应大于50%。
I . 如果GFP效率符合预期,即可以进入到后续的传代或筛选的步骤。
J . 如果G1/DR、G2/DR细胞GFP的荧光率在20~40%建议在转染后的48小时进行药物puro药物筛选;G1/DR、G2/DR荧光率在41~80%的可以按照传代后再进行药物筛选。(具体实验表面,电转后48小时开始药筛,对于GFP荧光率低的细胞具有更好的效果)
注意:
1)转染的过程使用的所有的培养基均为无抗生素培养基,否则将导致细胞死亡。
2)由于G1/G2载体使用的EF1a驱动GFP:P2A:Puro (如果增殖速度慢的细胞会出现GFP不强的情况)
3)如果G1/DR、G2/DR荧光率低于20%,建议重新优化转染实验条件。

图 6 K562细胞电转GFP效果图
3. 传代步骤
转染48h后,细胞按照1:5~1:10传代,培养3天后,再以1:5~1:10传代继续培养1周。(如果荧光率低,建议在转染后48小时直接开始药筛,以便后续的单克隆实验)
Note:细胞在第一次传代时,取混合的细胞株部分细胞进行PCR验证,判断混合细胞株是否有插入的敲除序列:具体参考第10步操作。
注意:由于Donor DNA载体中的嘌呤霉素抗性基因携带EF1a启动子,所以在基因组整合前的Donor DNA也会产生嘌呤霉素抗性。连续传代是为了稀释含有非整合型Donor DNA的细胞。嘌呤霉素筛选前的细胞传代时间越短,瞬时表达的假阳性率越高。
稀释传代的目的就是去除细胞核中游离的Donor DNA的抗性背景。
4. 药物筛选
细胞传代2~4次后,使用Puromycin(Puro)筛选或者GFP荧光分选。Puro的剂量需要通过杀伤曲线来确定,以找出药筛4-7天完全杀伤非转染细胞的最低剂量;嘌呤霉素的药筛范围为1 ~ 10 μg/ml),常规推荐使用浓度约为2 ug/ml,具体按照野生型细胞3~5天全部死亡最低浓度为准。
备注:经过药物筛选之后的携带有Puro抗性的细胞为两种基因型,随机插入基因型和定点插入导致敲除的基因型(目的基因型)。而鉴定的方式就是通过基因组的PCR进行鉴定。
Note:细胞进行单克隆之前,先取混合的细胞株进行PCR验证,初略判断混合细胞株插入的敲除序列丰度:具体参考第10步操作。
5. 细胞单克隆:药筛结束后,可采用下述三种方法进行单克隆化筛选细胞。
5.1. 极限稀释法:按照稀释后每个孔2~3个细胞进行稀释接种到96孔板中,适用于大部分生长状态良好的贴壁细胞和悬浮细胞。
根据细胞形成克隆的能力,确定96孔板单孔的接种数量,接种板数,将细胞按照一定的个数接种至96孔板中(具体依据细胞决定,通常在2~6个细胞,具体详见附录一)。
备注:此方法的克隆形成一般为14-21天,如遇21天仍未形成较为可观的克隆,进行正式敲除实验之前建议对使用的细胞进行摸索,或者询问购买的细胞库索要相关的参数。
5.2. 克隆环法:按照5% 的汇合度接种细胞,让细胞形成完整的独立的单克隆团,使用克隆环进行消化。
5.3. 流式筛选法:通过流式分选的方式,将带有GFP荧光的细胞分选到每个孔中。
5.4. 单克隆检测的数量一般在2x96个,具体依据实际情况调整。
6. 单克隆细胞PCR鉴定:96孔板传代(适用于贴壁细胞)
A. 吸去培养基,吸液枪头要放于孔侧壁,不与孔底面(细胞层)直接接触。
B. 加入100 μL/孔PBS进行清洗,吸去PBS,每孔加入30 μL的0.25%胰酶(胰酶提前预热至37℃),并将细胞放置于培养箱中消化作用。
备注:96孔板内的克隆,因生长时间较长,克隆也较为致密,一般需要更长的消化时间。消化过程中需选择一两个孔配合着吹打方式,观察细胞脱壁和消化的情况。
C. 消化完毕,加入50 μL培养基终止消化。
D. 用排枪充分吹打细胞,并按1:3的比例进行分板。取60 μL(3/4)的细胞悬液接种至新板,该板即为送检板(PCR检测板),原板内还残留20 μL(1/4)的细胞悬液,即为保留板(培养板)。
备注:传代比例可视具体情况进行调整。给予送检板更多的细胞是基于PCR需要一定的细胞数方能鉴定出来的原因。鉴定期间,保留培养板需要正常生长4-5d,才能拿到PCR结果。因此,传代比例需根据细胞的生长状态进行调整,生长较慢的细胞,需要接更多的细胞。
E. 为“保留板”和“送检板”分别补充130 μL和90 μL的培养基,并注意保留板四周孔需额外添加50 μL的培养基,防止培养基干掉。
F. 接种细胞的96孔板,放置在37°C、5%CO2、95%相对湿度的培养箱中培养,注意标记细胞代次。
G. 等待2~3天就可以进行PCR检测。
悬浮细胞:直接吸出60~70%的细胞悬液检测,并添加适量的培养基即可。
7. 单克隆细胞PCR鉴定
分离96孔板中培养中的单克隆细胞,待大部分送检PCR的96孔板里面的细胞汇合度达到70%~90%就可以进行DNA基因组提取。
8. 送检PCR前处理
贴壁细胞:
A. 送检96孔板内70%以上的孔,细胞汇合度能达70%~90%以上即可达到送检标准。
B. 吸去培养基加入200 ul PBS清洗一次,吸去PBS。
C. 进入基因组提取环节。
悬浮细胞:
A. 从原孔内吸取70%的细胞悬液,转移至96孔PCR板内(每孔200 ul容量,尖底),封上橡胶盖。
B. 样品需要使用离心机预处理,去除上清。
C. 进入基因组提取环节。
备注:如遇不能及时提取基因组的情况,需将96孔板置于-20℃保存。
9. 提取基因组DNA(按照使用基因组提取试剂盒进行提取操作)。
10. 进行PCR鉴定
Donor DNA(DR)的整合可能是正向(HDR)和反向(NHEJ)、发生切割产生indels。所以需要根据gRNA位点,设计鉴定引物,鉴定策略如下图:

图 8 正向/反向插入DR
表 3 基因鉴定引物的结果分析

A. 纯合子基因型:等位基因Donor DNA插入;
B. 纯合子基因型:等位基因Donor DNA插入、等位基因移码编辑;
C. 纯合子基因型:等位基因移码编辑;
D. 杂合子基因型:等位基因Donor DNA插入、等位基因未编辑;
E. 杂合子基因型:等位基因移码编辑、等位基因未编辑;
F. 野生型基因型:等位基因未编辑;
备注:由于细胞株不一定是二倍体,大部分是多倍体,所以具体的基因型要按照实际情况分析。
如图9,3.2 kb条带代表Donor DNA插入;0.5 kb条带代表未编辑或者存在indels (插入和删除)。对2种条带分别回收测序,分析基因型。

图 9 PCR鉴定与分析
11. 克隆扩增与实验应用
将鉴定正确的阳性单克隆细胞(纯合子/杂合子),继续扩大培养到足够细胞量冻存,或者直接用于下游实验。
——End——
附录:
(一)单克隆步骤确定每个Tip内的细胞个数方法如下:
A. 准备工作:暂定每孔添加的细胞悬液为10 μL每孔,即一个Tip的体积,向加样槽添加M mL培养基(10 μL*100孔*M块板)
B. 确定每孔接种细胞量为N个,接种板数为M块。
C. 计算可得需要取用的总细胞量为100NM(每孔N个细胞*100孔*M块板)
D. 重悬细胞沉淀并计数,得出细胞密度为ρ,计算需取用的细胞悬液体积为。
E. 吸取上述体积中的细胞,加入加样槽的培养基中吹打混匀。取2个10 μL上述体系,分别加入血球计数板的上下窗口,并于镜下观察计数整个10 μL镜下体系内的细胞数目(图示蓝色区域)。
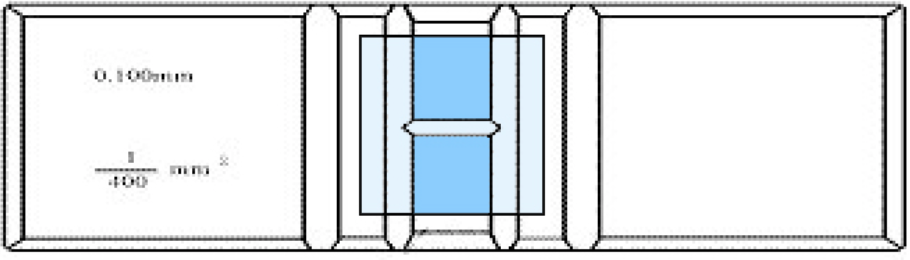
图 7 细胞计数板确认tip中细胞数量
备注:
* 计数时仅计数活细胞数量,死细胞不计入内;
* 上下两个窗口计数的平均值作为每个Tip的实际细胞数,记为N’;
* 若上下窗口计数相差较大甚至呈倍数相关(如2和6),考虑为加样槽细胞未混匀,需重新混匀并取样计数。
F. 根据预估每孔接种细胞量N与实际每Tip所含细胞量N’之间的差异,进行校正:
* 如N=N’±0.5~1,则每Tip体积:10 μL;
* 如N’-N>1,则每Tip体积:10 (N/N')μL;
* 如N-N’>1,则加样槽需再补加 10^5*M(N-N')/密度,原细胞悬液,重新混匀并进行步骤E操作。
G. 根据推算出的实际每Tip细胞悬液的体积,使用排枪对96孔板进行接种操作。
H. 标记好孔板信息,放置于37℃,5%CO2培养箱进行培养。